
Corso
BIOINFORMATICA


Corso
BIOINFORMATICA
|
|
AA 2024/2025 (in fase di aggiornamento) Docenti: Alessio Bechini (titolare) Codice: 688II CFU: 6 
La Bioinformatica è un campo multidisciplinare in cui si utilizzano gli strumenti propri dell'informatica per affrontare problematiche tipiche della biologia molecolare. In particolare, la Bioinformatica fa ricorso a tecniche di tipo computazionale per la gestione e l'analisi di dati biologici. Il corso fornisce un'introduzione ad alcune tematiche della
Bioinformatica, e in esso si enfatizzano i concetti e gli
strumenti informatici necessari per affrontare la
complessità dei sistemi analizzati. L'approccio seguito è
essenzialmente operativo: per la risoluzione dei problemi,
si prevede l'utilizzo congiunto di varie applicazioni
software e/o di un linguaggio di scripting. Gli argomenti
affrontati includono l'analisi di sequenze, gli studi
mutazionali e l'evoluzione molecolare, l'interazione con
banche dati biologiche, la modellazione molecolare, lo
studio strutturale delle proteine e le interazioni
molecolari nella cellula. Ove necessario, sono sviluppati
concetti basilari dell'informatica e dell'analisi numerica,
quali ad esempio la complessità computazionale e gli schemi
di integrazione. Il corso si prefigge lo scopo di sviluppare adeguatamente le capacità dello studente di Ingegneria Biomedica in campo informatico; al termine del corso lo studente dovrà aver acquisito le competenze necessarie per partecipare alla progettazione, implementazione e integrazione di sistemi software di tipo eterogeneo nel settore della Bioinformatica e della Biologia Computazionale. Prerequisiti: conoscenze dei fondamenti dell’informatica e padronanza di un linguaggio di programmazione; nozioni elementari di chimica, fisica e biologia molecolare.
- Si ricorda che le iscrizioni alla prova orale devono essere fatte obbligatoriamente tramite il portale unico di ateneo. - Le date di esame possono essere recuperate sul sito della Scuola di Ingegneria, e sono riportate sul Google Cal del corso (v. sez. "Orario").
-
Gli studenti che devono
sviluppare il progetto per l'esame sono invitati a contattare il
docente per concordare argomento e modalità di svolgimento. Prima lezione: Lun. 23 settembre 2024 Lun. 8:30-11:30 aula: C21; Giov. 14:00-15:45 aula: SI7. L'orario aggiornato delle lezioni è disponibile su un Google Cal pubblico, che può essere importato con il seguente indirizzo ICS: Il programma svolto è riportato nel registro delle lezioni; gli argomenti caratterizzanti sono indicati di seguito (in inglese) in forma categorizzata. La prova d’esame prevede due passi distinti:
1) Svolgimento di un piccolo progetto con sviluppo di software
in Python, da svolgersi in 1-3 persone, su una tematica concordata
con il docente. Si sottolinea esplicitamente che, nelle prove d'esame, il candidato viene valutato anche in relazione alla sua capacità di esprimersi in linguaggio tecnico appropriato. Il materiale didattico del corso è riportato sul MS Teams a cura del docente. Tutti coloro che abbiano bisogno di un compendio delle nozioni basilari di biologia molecolare richieste per seguire al meglio il corso, possono far riferimento a: Lawrence Hunter, Molecular biology for computer scientists ch. in Artificial intelligence and molecular biology (1993): 1-46. Testi (per approfondimenti):  T1
-
Titolo Python for Biologists
T1
-
Titolo Python for BiologistsAutore Martin Jones Editore CreateSpace Independent Publishing Platform; 1° edizione (7 settembre 2013) ISBN 978-1492346135 Note Testo con riferimenti a problemi di tipo biologico  T2
-
Titolo Fondamenti di bioinformatica
T2
-
Titolo Fondamenti di bioinformaticaAutori M. Citterich, F.Ferrè, G. Pavesi, C. Romualdi, G.Pesole Editore Zanichelli ISBN 9788808621122 Note Testo con molti dettagli di tipo biologico (solo parzialmente coperti in questo corso) Esercitazioni: La versione di Python utilizzata nel corso è la 3.x, ma si presentano anche le caratteristiche della 2.7. L'ambiente di sviluppo di riferimento è Spyder. Si consiglia di utilizzare la versione integrata nella distribuzione Anaconda (v. sezione "LINKS"). Miscellanea: G1 - glossario di alcuni termini bionformatici (portale ROSALIND) Seq0 - Sequenze: DNA mitocondriale umano (FASTA) L1 - pythontutor.com - sito web interattivo per la visualizzazione dell'esecuzione di codice Python C0 - Implementazione Python di Needleman-Wunsch + esempi - allineamento_globale.zip M0 - asn.pdb asparagina - gly.pdb glicina - phe.pdb fenilalanina - pro.pdb prolina M1 - 1hho.pdb emoglobina (oxi) - 2hhb.pdb emoglobina (deoxi) L3 - animazione con esempio di modi normali di vibrazione molecolare
Una lista degli strumenti software più utilizzati nel settore della proteomica si può trovare sul sito di ExPASy. Chi è interessato a impieghi nel settore della bioinformatica e della biologia computazionale può consultare le pagine dedicate a cura della BITS, di ISBC e di bioinformatics.org I dettagli riguardanti lo svolgimento di ciascun progetto devono essere concordati con il docente. A.A. 2014/2015 - 12/06/2015 03/07/2015 24/07/2015 11/09/2015 15/01/2016 04/02/2016 23/02/2016 A.A. 2015/2016 - 08/06/2016 29/06/2016 20/07/2016 12/09/2016 11/01/2017 31/01/2017 20/02/2017 A.A. 2016/2017 - 08/06/2017 29/06/2017 20/07/2017 20/09/2017 11/01/2018 01/02/2018 19/02/2018 A.A. 2017/2018 - 07/06/2018 28/06/2018 13/09/2018 10/01/2019 31/01/2019 18/02/2019 A.A. 2018/2019 - 06/06/2019 18/07/2019 17/09/2019 10/01/2020 30/01/2020 20/02/2020 Curiosità
I link in questa sezione si riferiscono a iniziative inerenti gli argomenti trattati nel corso. Animazione algoritmo di
Nussinov - visualizzazione didattica sviluppata come
progettino di esame (Favale/Vivaldi); versione .exe
Note
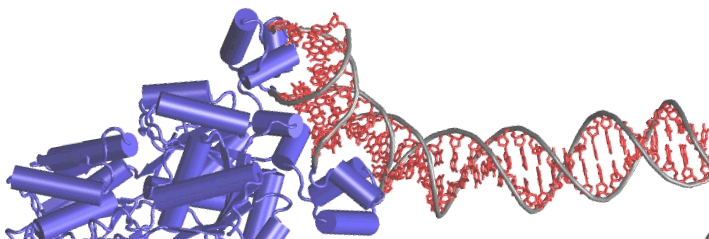
L'immagine nella parte destra dell'intestazione mostra un particolare dell'aggancio sul DNA della proteina "lac repressor", con funzioni di regolazione genetica in E. Coli (TCB - Univ. of Illinois at Urbana-Champain) L'immagine di sfondo nella colonna del menù a sinistra riporta una porzione di un modello dell'insulina ottenuto con il software VMD. L'immagine nella sezione della descrizione del corso è un "tag cloud" di alcuni termini ricorrenti in articoli di bioinformatica. |