
Corso
STRUMENTI
SOFTWARE
PER LA BIOLOGIA COMPUTAZIONALE


Corso
STRUMENTI
SOFTWARE
PER LA BIOLOGIA COMPUTAZIONALE
| |
AA 2009/2010 Docente: Alessio Bechini Cod: II619 CFU: 3 ATTENZIONE: per l'a.a. 2009/2010 i contenuti del corso verranno svolti nell'ambito del corso di "Bioinformatica". Di conseguenza, chi sostiene l'esame di Bioinformatica con tale programma non pu˛ inserire "Strumenti software..." come corso a scelta. Viceversa, sebbene "Strumenti software..." non venga formalmente attivato, sarÓ comunque possibile seguire le lezioni con il programma relativo (il mercoledý pomeriggio) e successivamente sostenere regolarmente l'esame. Per ulteriori chiarimenti contattare il docente.  La Biologia Computazionale Ŕ un campo multidisciplinare in cui si sfruttano le tecniche tipiche dell'informatica per riuscire a comprendere i sistemi biologici a livello molecolare. Il corso fornisce un'introduzione ad alcune tematiche della Biologia Computazionale, e in esso si enfatizzano i concetti e gli strumenti informatici necessari per affrontare la complessitÓ dei sistemi analizzati. L'approccio seguito Ŕ essenzialmente operativo: per la risoluzione dei problemi, si prevede l'utilizzo congiunto di varie applicazioni software e/o di un linguaggio di scripting. Gli argomenti affrontati includono la modellazione molecolare, lo studio strutturale delle proteine e le interazioni molecolari nella cellula. Il corso si affianca idealmente a quello di Bioinformatica e si prefigge lo scopo di sviluppare adeguatamente le capacitÓ dello studente di Ingegneria Biomedica in campo informatico; al termine del corso lo studente dovrÓ aver acquisito le competenze necessarie per partecipare alla progettazione, implementazione e integrazione di sistemi software di tipo eterogeneo nel settore della Biologia Computazionale. Prerequisiti: conoscenze dei fondamenti dell’informatica e padronanza di un linguaggio di programmazione; nozioni elementari di chimica, fisica e biologia molecolare. -
Gli studenti che devono sostenere l'esame sono pregati di contattare il
docente per concordare le modalitÓ di svolgimento del progetto.
- Nel giorno di mercoledý 26 maggio si terra' lezione anche durante il mattino, dalle 9:00 alle 11:30 in aula SI6.
Il programma svolto Ŕ riportato nel registro delle lezioni; gli argomenti caratterizzanti sono indicati di seguito in forma categorizzata. La prova d’esame si svolge, di
norma, come segue : Si sottolinea esplicitamente che, nella prove d'esame, il candidato viene valutato anche in relazione alla sua capacitÓ di esprimersi in linguaggio tecnico appropriato. La lista per le prove viene compilata prima
dell'inizio delle stesse a cura degli studenti. Il materiale didattico consigliato verrÓ riportato in questa sezione. Testi:  T1
- Titolo
Python in a Nutshell, 2nd ed.
T1
- Titolo
Python in a Nutshell, 2nd ed.Autore Alex Martelli Editore O'Reilly ISBN 0-596-10046-9 Miscellanea: M0 - asn.pdb asparagina - gly.pdb glicina - phe.pdb fenilalanina - pro.pdb prolina M1 - 1hho.pdb emoglobina (oxi) - 2hhb.pdb emoglobina (deoxi) L1 - pagina web con nozioni di base di molecular mechanics L2 - animazione con esempio di modi normali di vibrazione molecolare S1 - slide su espressioni regolari in Python (sito Dalke Scientific Software) T1 - tutorial sulla libreria matplotlib D1 - descrizione dei formati dei file usati da NAMD
Una lista degli strumenti software pi¨ utilizzati nel settore della proteomica si pu˛ trovare sul sito di ExPASy. Chi Ŕ interessato a impieghi nel settore della biologia computazionale pu˛ consultare le pagine dedicate a cura della BITS, di ISBC e di bioinformatics.org Di seguito si riportano alcune possibili argomenti da trattare nei progetti. I dettagli riguardanti lo svolgimento di ciascun progetto devono essere concordati con il docente.
CuriositÓ
I link in questa sezione si riferiscono a iniziative inerenti gli argomenti trattati nel corso.
Note
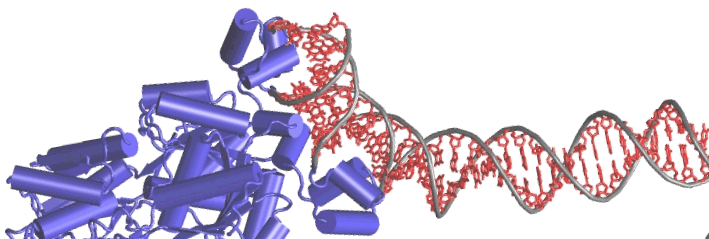
L'immagine nella parte destra dell'intestazione mostra un particolare dell'aggancio sul DNA della proteina "lac repressor", con funzioni di regolazione genetica in E. Coli (TCB - Univ. of Illinois at Urbana-Champain) L'immagine di sfondo nella colonna del men¨ a sinistra riporta una porzione di un modello dell'insulina ottenuto con il software VMD. L'immagine nella sezione della descrizione del corso Ŕ una porzione di grafo ottenuto con Cytoscape per la visualizzazione e l'analisi del transcriptoma. |